Étude de la végétation
L’index de végétation est une valeur calculée, déterminant l’état d’une plante dans son cycle de vie. Ce ratio résulte de l’observation de longueurs d’ondes émises par celle-ci. En effet, les arbres, le sol, les forêts émettent des longueurs d’ondes (des couleurs) que nos yeux peuvent capter ou non. L’index de végétation, couramment nommé « NDVI » — pour « Normalised Difference Vegetation Index », désigne la différence d’émission entre les couleurs rouge et le proche infra rouge, en anglais « NIR ».
Le ratio sera donc le suivant, similaire à un calcul de contraste.
NIR - Rouge
NDVI = ------------
NIR + Rouge
On parle bien ici d’intensité de couleur. Les valeurs de ce ratio déterminent, en pratique, les types de sols suivants:
- NDVI = [0.6, 1] : végétation dense
- NDVI = [0.2, 0.5] : champs agricoles
- NDVI ~ 0 : rochers, neige, sable
- NDVI ~ -1 : eau
Dans la mesure où l’on peut donc détecter les différents types de végétation, les chercheurs peuvent donc étudier l’évolution des forêts, des champs,… Mais comment récupérer ces intensités de couleurs ?
Les satellites artificiels en orbites font des photographies par zones de différentes longueurs d’ondes. Et entre autres, les longueurs d’ondes rouge et infrarouge, qui nous intéressent. Pour calculer le NDVI il faut donc récupérer les images satellites traitant d’une zone donnée, en extraire les bandes NIR et rouge et appliquer le calcul de contraste pixel par pixel. Une nouvelle image sera créée, comme une carte, comportant une seule valeur par pixel.
Certaines organisations proposent des cartes NDVI précalculées. Il est alors aisé pour le scientifique de les télécharger, et ensuite de les projeter sur une carte, ou une image satellite réelle.
Télécharger les données
L’entreprise Londonienne AgroMonitoring propose des APIs de récupération de cartes NDVI. Elle propose une souscription gratuite ou payante selon la localité ou la résolution.

def get_final_images(self, polygon_id:str, start_date:datetime.datetime, end_date:datetim.datetime) -> bool:
"""get NDVI rasters of a polygon id during the interval between two dates,
and map them on a satellite image
"""
# call a dedicated method to retrieve upper and lower bounds
ullng, ullat, lrlng, lrlat = self._getPolygonCoords(polygon_id)
start_ts = round(start_date.timestamp())
end_ts = round(end_date.timestamp())
d1 = start.strftime("%d-%m-%Y")
d2 = end.strftime("%d-%m-%Y")
print(f"Retrieving NDVI images from {d1} to {d2}")
# generate the query
params = {
"start": start_ts,
"end": end_ts,
"polygon_id": polygon_id,
"appid": self._apitoken,
"resolution_min": 10
}
url = f"https://api.agromonitoring.com/agro/1.0/image/search"
try:
result = requests.get(url, params=params).json()
except Exception as e:
print(f"Could not retrieve NDVI image: {e}")
return False
if not result:
print("No NDVI image found...")
return False
rlen = len(result)
print(f"Found {rlen} NDVI images")
for res in result:
acq_ts = int(res["dt"])
acq_time = datetime.datetime.utcfromtimestamp(acq_ts).strftime('%Y-%m-%d %H:%M:%S')
sat_type = res["type"].replace(" ","_")
if "landsat" in sat_type.lower():
# skip it
continue
ndvi_stored_path = f"image_{polygon_id}_{acq_ts}_{sat_type}_ndvi.png"
if not os.path.isfile(ndvi_stored_path):
print(f"NDVI image {ndvi_stored_path} not found on disk, downloading")
res = requests.get(res["image"]["ndvi"])
try:
f = open(ndvi_stored_path, "wb")
f.write(res.content)
except Exception as e:
print(f"Could not write image to disk {ndvi_stored_path}: {e}")
return False
finally:
f.close()
# now call a R script that maps the NDVI image on top of a satellite image
ndvi_projected_path = f"image_{polygon_id}_{acq_ts}_{sat_type}_ndvi_projected.png"
try:
self.call_r_routine(ndvi_stored_path, ndvi_projected_path, ullng, ullat, lrlng, lrlat)
except Exception as e:
print(f"Could not execute the images mapping: {e}")
return False
else:
return True
Le lien entre Python et R
L’utilisation de bibliothèques telles que Reticulate sont intéressantes pour l’appel de script Python dans la session R. Dans le sens inverse qui nous intéresse présentement, le module rpy2 répond à cette problématique d’appeler un script R dans la session Python.
Mais dans ce contexte d’expérimentation, les exigences techniques ne sont pas les mêmes que pour un logiciel final à utiliser fréquemment. On se limitera ici à forker un processus sur une machine GNU/Linux.
def call_r_routine(self, infilename:str, outfilename:str, ullng:float, ullat:float, lrlng:float, lrlat:float):
"""call a R script"""
command = f"./r_process_images.R {infilename} {outfilename} {ullng} {ullat} {lrlng} {lrlat}"
try:
process = subprocess.Popen(command, shell=True, stdout=subprocess.PIPE, stderr=subprocess.STDOUT)
except Exception as e:
raise e
output = process.communicate()[0]
exitCode = process.returncode
if exitCode == 0:
return output
else:
raise RuntimeError
Superposer les images
Voici le script R, qui gère le système de coordonnées, télécharge l’image satellite et calque les deux images entre elles.
#!/usr/bin/Rscript
library(raster)
library(RColorBrewer)
library(OpenStreetMap)
library(png)
args = commandArgs(trailingOnly=TRUE)
if (length(args) < 6){
stop()
}
infilename <- args[1]
outfilename <- args[2]
ullng <- as.numeric(args[3])
ullat <- as.numeric(args[4])
lrlng <- as.numeric(args[5])
lrlat <- as.numeric(args[6])
# load the image as a raster
r <- raster(infilename)
# set the colors palette
cuts <- seq(-1, 1, 0.1) #set breaks
create_linear_palette <- function(x){
pal <- colorRampPalette(c("#0000FF", "#FF0000", "#FFFF00", "#00FF00"))
return(pal(x))
}
# polygon coordinates
ul <- c(ullat, ullng)
lr <- c(lrlat, lrlng)
ROI <- c(ullng, lrlng, lrlat, ullat)
# set extent
ext <- extent(ROI)
extent(r) <- ext
values(r) <- (values(r)/125)-1
# get map
mmap <- openmap(ul, lr, zoom=19)
crs(r) <- "+proj=longlat +datum=WGS84 +no_defs +ellps=WGS84 +towgs84=0,0,0"
# project to map
CRS <- crs(raster(mmap))
r.proj <- projectRaster(r, raster(mmap), crs(CRS))
# plot
png(file = outfilename, width = 800, height = 700)
plot(mmap)
plot(r.proj, alpha=0.5, add=T, col=create_linear_palette(20), breaks=cuts)
dev.off()
Résultat
Regardons d’abord le lieu que nous souhaitons analyser. Il s’agit d’un village de montagne. Voici l’image satellite.

Et voici la carte open source.
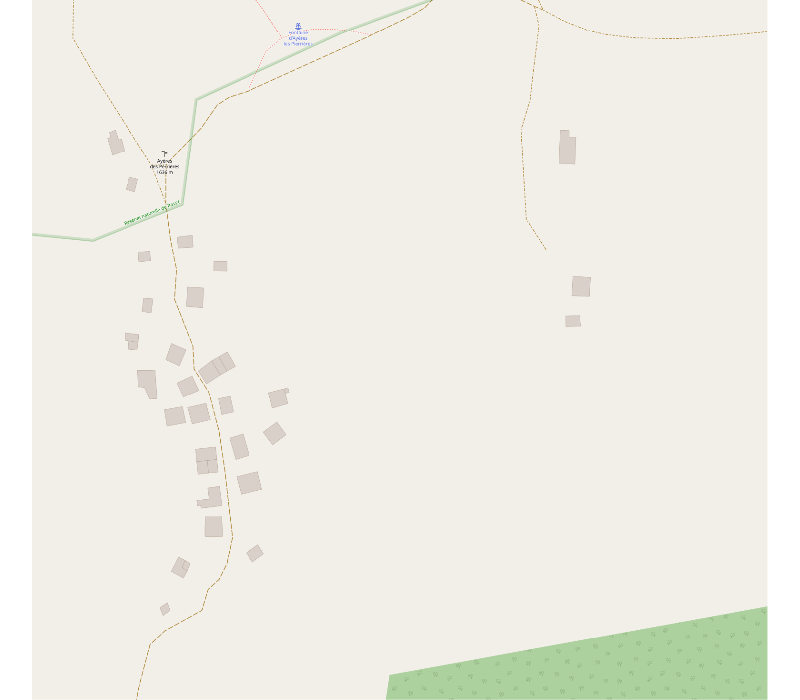
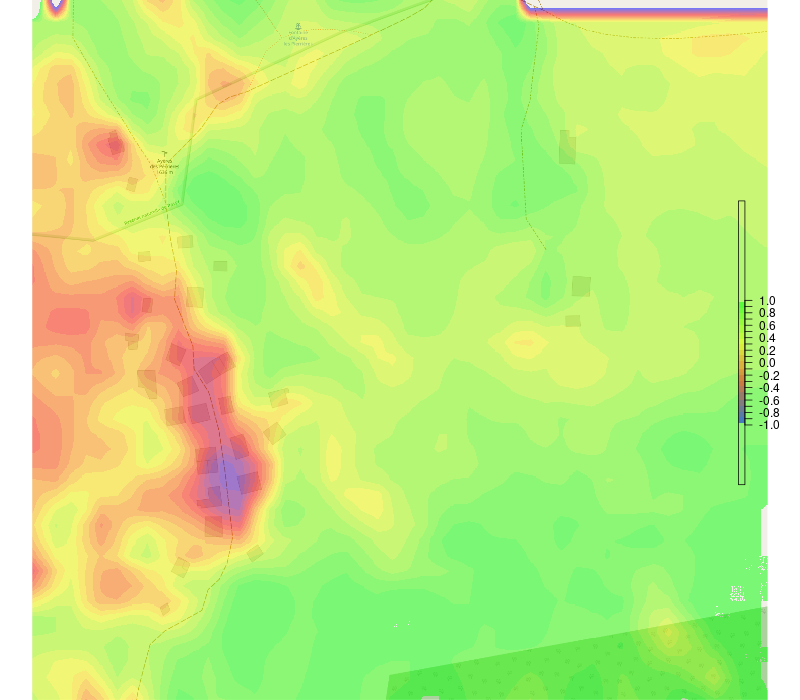
Pour terminer on superpose la carte NDVI sur la carte open source, avec une image du NDVI d’une résolution d’un pixel pour 10 mètres. On note bien les différences de niveau de l’indicateur NDVI ; notamment sur la partie de gauche où se situent les habitations.